El nanomundo en tus manos
7. Nanobiotecnología y nanomedicina
Página 17 de 22
— 7 —
NANOBIOTECNOLOGÍA Y NANOMEDICINA
Una de las cuestiones que más me atraían era la estructura del cuerpo humano y de cualquier ser vivo. Frecuentemente me preguntaba de dónde vendría el principio de la vida. Era una pregunta osada, ya que siempre se ha considerado un misterio. Tras noches y días de increíble labor y fatiga, conseguí descubrir el origen de la generación, de la vida; es más, yo mismo estaba capacitado para infundir vida en la materia inerte. Me preparé para múltiples contratiempos; mis intentos podrían frustrarse, y mi labor resultar finalmente imperfecta. Sin embargo, me animaba cuando consideraba los avances que día a día se producen en las ciencias y la mecánica, pensando que mis experimentos al menos servirían como base para futuros éxitos. Tampoco podía tomar la amplitud y complejidad de mi proyecto como excusa para no intentarlo siquiera. Imbuido de estos sentimientos, comencé la creación de un ser humano.
MARY W. SHELLEY, Frankenstein o el moderno Prometeo, 1818
7.1. La vida y el nanomundo
Decenas de millones de especies habitan en nuestro planeta. En todas ellas, tanto en los omnipresentes microorganismos como en los seres pluricelulares, su unidad estructural y funcional es la célula. Sin embargo, las células son sistemas relativamente complejos y muy organizados, y dentro de ellas bulle un mundo de moléculas reaccionando e interaccionando entre sí, formando agregados de diferente tipo que se sintetizan y degradan constantemente. La vida es, en el fondo, una constante danza de moléculas dominadas por los mismos parámetros termodinámicos y cinéticos que rigen toda la química, con la particularidad de que dichas moléculas y sus interacciones han sido seleccionadas por la evolución. Para ello, la vida requiere la existencia de compartimentos celulares en los que la materia se ordena gracias a un continuo consumo de energía, frente a su heterogéneo y cada vez más desordenado entorno. El consumo de energía por el sistema vivo le permite mantenerse alejado del equilibrio termodinámico y ser capaz de evolucionar. De hecho, no existe vida sin evolución, como ya Charles R. Darwin demostró hace un siglo y medio.
Las biomoléculas que nos constituyen son nanoestructuras, disposiciones tridimensionales de átomos cuyo tamaño está en el rango de los nanómetros. Durante las últimas décadas hemos avanzado mucho en el estudio de la estructura y función de las moléculas biológicas, y las herramientas de la nanotecnología nos permiten analizarlas y manipularlas una a una. Así, un nuevo campo, conocido como «nanobiotecnología» o «bionanotecnología» ha surgido con fuerza gracias al esfuerzo combinado de biólogos, químicos, físicos e ingenieros. Éste es un claro ejemplo de la convergencia de ciencias de la cual hablaremos en el Capítulo 8. Con ello, el mundo de lo «nanobio» está ya cambiando la biotecnología y la medicina, y sus avances van a condicionar desde los biosensores que usamos hasta los fármacos que puedan salvarnos la vida. Además, estos desarrollos nos están permitiendo comprender mejor cómo pudo originarse la vida, con lo que también nos acercan a una posibilidad aún remota pero ciertamente inquietante: sintetizar seres vivos en el laboratorio ensamblando sus moléculas una a una.
7.2. La química de la vida
Los seres vivos estamos constituidos por un número muy limitado de elementos de la tabla periódica. De hecho, sólo seis de ellos son responsables del 99% de la masa de casi cualquier organismo: oxígeno (cuyo símbolo en la tabla periódica es O: de media, el 65,9% de la masa), carbono (C: 18,3%), hidrógeno (H: 10,2%), nitrógeno (N: 3,2%), fósforo (P: 1,1%) y azufre (S: 0,3%). El 1% restante lo forman los conocidos como «oligoelementos», entre los que están (empleando su símbolo): Na, K, Mg, Ca, Cr, Mo, Mn, Fe, Co, Ni, Cu, Zn, B, Al, Si, Sn, Se, F, Cl y I. La abundancia del oxígeno y el hidrógeno se debe a que el agua (H2O) es un componente fundamental de los seres vivos, en los que actúa como soporte estructural, estabilizador térmico y disolvente, y en cuyo seno se realizan la mayor parte de las reacciones químicas que requiere la vida. Además, la alta proporción de carbono nos dice que ése es el elemento fundamental en las biomoléculas, ya que posee unas propiedades inmejorables para formar enlaces covalentes consigo mismo y con los demás elementos mayoritarios de la vida. Por tanto, somos básicamente agua y carbono.
El agua, las sales minerales y las moléculas más sencillas de los seres vivos (como el NH3, el NO, o el CO2), poseen un tamaño de tan sólo 0,2-0,4 nm. El siguiente grupo (en complejidad y tamaño) lo forma un amplio repertorio de compuestos orgánicos pequeños como glicerol, urea, glucosa, vitaminas, algunas hormonas y diferentes tipos de biomoléculas que son transformados constantemente por el metabolismo, y tienen tamaños en el rango de 0,3 a 0,6 nm. Entre estas moléculas pequeñas están también los monómeros de las macromoléculas biológicas: ácidos grasos (por ejemplo, el ácido oleico o el palmítico), azúcares simples monosacáridos (como la glucosa o la fructosa), aminoácidos (entre ellos los veinte que constituyen las proteínas), y nucleótidos (cinco de los cuales forman los ácidos nucleicos, y algunos como el adenosín trifosfato o ATP son además moléculas ricas en energía usadas en el metabolismo). Su tamaño está entre 0,4 y 1 nm. Por último, las macromoléculas biológicas son los lípidos complejos (como los fosfolípidos que forman las membranas celulares), los polisacáridos (por ejemplo, la celulosa), las proteínas, y los ácidos nucleicos (ácido desoxirribonucleico o ADN y ácido ribonucleico o ARN). Algunos tamaños típicos de las macromoléculas son los siguientes: diámetro de las hélices que pueden formar los aminoácidos al unirse, 0,46 nm; diámetro de la doble hélice del ADN, 2 nm; diámetro de la hemoglobina (proteína que transporta el oxígeno en nuestra sangre), 6,4 nm; grosor de la membrana celular: 7,5 nm. En la Figura 7.1 se muestran ejemplos de biomoléculas.

FIGURA 7.1. A. Ejemplos de moléculas biológicas: un azúcar (glucosa), un ácido graso (ácido palmítico), un aminoácido (alanina) y un nucleótido (adenosín trifosfato o ATP). B. Esquema del flujo de información genética en los seres vivos: el paso de ADN a ARN se denomina transcripción, y el de ARN a proteínas traducción; algunos virus (denominados «retrovirus») son capaces de realizar una retrotranscripción de ARN a ADN.
Proteínas y ácidos nucleicos son las macromoléculas fundamentales y más características de la vida. Las proteínas pueden desempeñar diferentes funciones, entre ellas catalizar reacciones metabólicas (las denominadas «enzimas»), actuar como transportadores específicos a través de la membrana celular, servir de armazón estructural en la célula, unirse a azúcares o lípidos, o formar distintos tipos de agregados macromoleculares con los ácidos nucleicos. Por su parte, los ácidos nucleicos son los biopolímeros que se encargan de almacenar y transmitir la información hereditaria de los seres vivos, ya que constituyen su genoma, es decir, el conjunto de la información genética que posee una especie. Los genomas de todos los organismos celulares son de ADN de cadena doble (dsADN), mientras que en el caso de los virus, de los que hablaremos en el apartado 7.4, sus genomas pueden ser de ADN o de ARN, y tanto de cadena doble (dsADN y dsARN) como sencilla (ssADN y ssARN). La investigación en biología molecular ha permitido demostrar que el flujo de información genética entre las macromoléculas fundamentales de la vida sigue el esquema ADN ↔ ARN → proteína (Figura 7.1B).
7.3. Donde el microscopio no llega
Durante mucho tiempo se sospechó que muchos fenómenos naturales y artificiales estaban producidos por algún tipo de criaturas vivas desconocidas, que resultaban demasiado pequeñas como para ser observadas a simple vista (los humanos no podemos ver objetos que midan menos de unos 50 μm, aproximadamente el grosor de un cabello) o incluso utilizando una lupa. Algunos de esos procesos eran muy útiles para la humanidad, como la fermentación del pan o la cerveza (llevada a cabo por ciertas levaduras) y otros eran tan destructivos como la lepra o la peste (producidas, respectivamente, por las bacterias Mycobacterium leprae y Yersinia pestis). Tales microorganismos misteriosos pudieron observarse por primera vez a finales del siglo XVI, con la invención del microscopio de manera casi simultánea por G. Galilei en Italia y Z. Janssen en Holanda. Los primeros microscopios eran instrumentos muy simples formados por un par de lentes alineadas, una cercana al objeto que se pretendía observar (llamada por ello «objetivo») y otra cercana al ojo del observador (el «ocular»), separadas entre sí por un tubo hueco de unos 8 o 10 cm. Con ellos era posible aumentar la imagen hasta unas 200 veces, aunque debido a lo rudimentario del proceso de fabricación y al mal pulido de las lentes, lo que se observaba aparecía generalmente borroso y poco definido.
Las primeras observaciones relevantes al microscopio se hicieron más de medio siglo después de su invención, cuando en 1664 R. Hooke observó una fina lámina de corcho y comprobó que era un material poroso formado por cajas o celdillas, para las que acuñó un nombre que pasaría a la historia: «células». Unos años más tarde, el biólogo M. Malpighi observó por primera vez al microscopio células vivas (de entre 10 y 20 μm de tamaño) formando parte de tejidos de animales y plantas. Más tarde, A. van Leeuwenhoek perfeccionó el dispositivo usando lentes más pequeñas y potentes, construidas con vidrio de mayor calidad y mucho mejor pulidas. Gracias a los casi 500 aumentos de su microscopio, alrededor del año 1676 logró observar por primera vez la gran cantidad de microorganismos, con colores y morfologías diferentes, que contenía el agua estancada. Además, quien hoy consideramos como padre de la microbiología dibujó con mucho cuidado lo que él llamaba «diminutos animálculos», y en 1683 llegó a observar incluso bacterias (cuyo tamaño es de entre 1 y 2 μm). A lo largo del siglo XVIII los microscopios se fueron perfeccionando en sus partes mecánicas, lo que permitió un aumento en su estabilidad y facilidad de uso.
Las mejoras relevantes en la óptica del microscopio se produjeron cuando en 1877 el físico E. K. Abbe publicó su teoría del microscopio y además mejoró la microscopía de inmersión tal como le había encargado el óptico y fabricante de microscopios C. Zeiss. Así, sustituyendo el agua por aceite de cedro se aumentaba el índice de refracción del medio de 1,33 a 1,5, lo que permitía lograr unos 1000 aumentos. Con ello era posible observar las bacterias con detalle (Figura 7.2A), y también distinguir el núcleo y orgánulos de las células de animales o plantas. En los años veinte casi se había alcanzado el límite teórico de aumento para los microscopios ópticos y se habían desarrollado distintos tipos de iluminación para resaltar el contraste de la muestra. No obstante, era evidente que la vida estaba constituida por componentes de tamaños menores, que era necesario observar y estudiar.
La siguiente revolución se produjo en los primeros años treinta, cuando los físicos E. Ruska y M. Knoll construyeron el primer microscopio electrónico. En él se hacía llegar a la muestra un haz de electrones en lugar de luz visible, con lo que se abría la posibilidad de observar estructuras intracelulares muy pequeñas, y microorganismos de distinto tipo incluyendo los virus. Eso sí, al no utilizar luz visible en realidad se dejaba de «ver» realmente el espécimen, y la imagen producida se debía a la reconstrucción de las señales producidas por la interacción de los electrones con la materia. Con ello, evidentemente, se perdía la información sobre el color de la muestra. No obstante, en ocasiones las imágenes en blanco y negro obtenidas por microscopía electrónica se representan con falsos colores para indicar las diferentes alturas de la muestra, realzar su contraste, o bien resaltar ciertas partes del objeto observado. Hoy en día, las microscopías electrónicas de transmisión (TEM) y de barrido (SEM, Figura 7.2B), descritas en el Capítulo 3, poseen una enorme potencia, por lo que permiten visualizar estructuras moleculares de dimensiones nanométricas como proteínas o ácidos nucleicos, e incluso átomos.

FIGURA 7.2. Imágenes de bacterias del género Bacillus observadas con un microscopio óptico (A), SEM (B) y AFM (C). Imágenes tomadas por A. I. López-Archilla (Universidad Autónoma de Madrid), M. Madhaiyan (Universidad Nacional Chungbuk, Corea del Sur) y E. López-Camacho (Centro de Astrobiología, CSIC-INTA).
No obstante, la microscopía electrónica posee una limitación fundamental en el ámbito de la biología: dado que la muestra observada no puede contener agua líquida, resulta imposible ver células vivas, y tampoco estructuras moleculares en disolución. Otra limitación es el hecho de que la materia orgánica, y en especial la biológica, se degrada rápidamente bajo la acción de los electrones. Además, dado que los haces de electrones tienen escaso poder de penetración, si se desea visualizar por microscopía TEM los componentes celulares, incluso una célula aislada es demasiado gruesa, y es preciso cortarla en láminas ultrafinas de unos 20-60 nm de espesor. Cuando usamos la microscopía TEM, para obtener suficiente contraste, las preparaciones se han de «teñir» previamente con compuestos que poseen átomos pesados (por ejemplo, sales de uranio, plomo o lantano). En el caso del SEM, el espécimen cuya superficie se desea observar ha de ser recubierto con una capa delgada de un material metálico, como el oro. No obstante, a pesar de estas limitaciones, los nuevos avances que se están incorporando tanto en la tecnología como en el procesamiento de las imágenes permiten predecir un largo y exitoso futuro de las microscopías electrónicas para estudiar los componentes de los seres vivos.
En cualquier caso, las microscopías electrónicas convencionales poseen un límite de resolución en el rango de los nanómetros a partir del cual no resultan útiles. Para estudiar los nanobjetos, que como estamos viendo son los protagonistas moleculares del mundo vivo, se han desarrollado algunas modificaciones experimentales de la microscopía electrónica, y además se han ideado tecnologías totalmente diferentes. Entre ellas están la difracción de rayos X, la resonancia magnética nuclear, los nuevos tipos de microscopía de campo cercano (STM y AFM, Figura 7.2 C), y otros sorprendentes avances experimentales, algunos de los cuales se han descrito en capítulos anteriores de este libro.
7.4. Virus y ribosomas: dos tipos de nanomáquinas naturales
Además de las células y los organismos pluricelulares formados por ellas, en la naturaleza existen otros tipos de «entidades replicativas» sobre las que (como veremos en el apartado 7.7) existe un debate científico acerca de si son seres vivos o no: los viroides y los virus. Los viroides son moléculas de ARN circular y «desnudo» (sin ningún tipo de cobertura proteica), de entre 240 y 400 nucleótidos de longitud, que infectan numerosas especies de plantas y pueden producirles enfermedades. Desde el punto de vista tecnológico, se ha propuesto que moléculas de este tipo podrían ser utilizadas como herramientas para inducir distintos tipos de respuestas en plantas, tanto per se como unidas a algún tipo de nanoestructura.
Por su parte, los virus son parásitos capaces de infectar a todas las especies celulares conocidas, y están formados por tres componentes principales: a) un genoma, que como se indicaba en el apartado 7.2 puede ser de ADN o de ARN; b) una cápsula o «cápsida» formada por proteínas, que protege al genoma; y c) en algunas familias virales, una membrana lipídica que ha sido «robada» a la última célula que han infectado. Los virus con genoma de ARN (como el de la gripe, el del sida o el del Ébola) evolucionan mucho más rápidamente que los que tienen genoma de ADN (como el de la varicela, el papiloma o el herpes) debido a que las enzimas que copian su genoma producen más errores o mutaciones. Por ello, estos virus generan rápidamente mutantes resistentes a los fármacos y es mucho más difícil luchar contra ellos cuando nos infectan. La existencia de los virus ya fue intuida por L. Pasteur a mediados del siglo XIX, cuando no pudo encontrar el microorganismo que producía una enfermedad entonces mortal, la rabia, y propuso que debería ser una entidad patógena diferente a los «microbios» o «toxinas» entonces conocidos: bacterias, hongos y protozoos. Además, el patógeno causante de la rabia debía ser muy pequeño, ya que era imposible observarlo al microscopio óptico. Por tanto se estaba luchando contra un enemigo invisible, hoy sabemos que de dimensiones nanométricas.
Los primeros virus fueron descubiertos a finales del siglo XIX, aunque hubo que esperar hasta la aparición de la microscopía electrónica para comenzar a visualizarlos, y con ello estudiar sus variadas morfologías y diferentes procesos de infección en las células. El uso de TEM y SEM, junto con otras técnicas estructurales como la difracción de rayos X, ha mostrado que el tamaño típico de los virus es de entre 20 y 300 nm (es decir, aproximadamente entre 3 y 50 veces más pequeños que las bacterias), si bien hay algunas excepciones de dimensiones mayores. Las morfologías de los virus son muy variadas. En la Figura 7.3 se muestran esquemas de la estructura de diversos virus, y algunas imágenes obtenidas por microscopía electrónica.
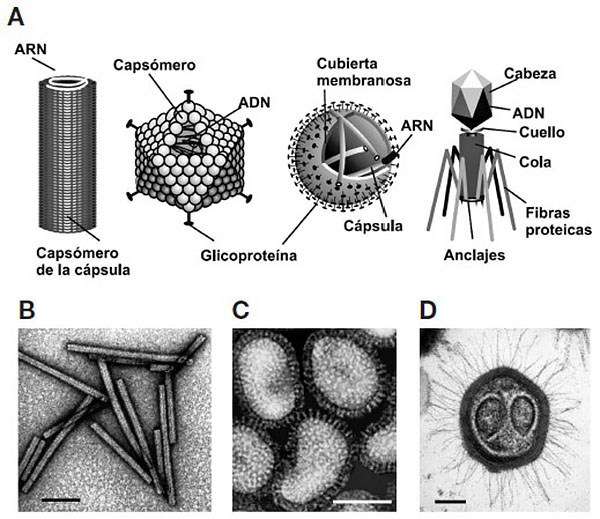
FIGURA 7.3. A. Representación esquemática de la estructura y componentes de distintos virus: helicoidal, icosaédrico, esférico, y un bacteriófago que infecta a bacterias. B. Imágenes TEM de tres especies de virus: el virus del mosaico del tabaco, el virus de la gripe, y el mimivirus (uno de los de mayor tamaño conocido). En los tres casos, la barra indica 100 nm. Procedencia de las figuras de TEM: B, H.-W. Ackermann (Universidad de Laval, Quebec, Canadá); C, L. Stannard (Universidad de Ciudad del Cabo, República de Sudáfrica); D, D. Raoult (Universidad del Mediterráneo, Francia).
Durante las últimas décadas también se han ido desvelando los mecanismos que emplean muchos de los virus conocidos para infectar a las células y poner a su servicio parte de la maquinaria replicativa y metabólica celular. Los ciclos infectivos virales siguen estrategias diferentes, unas muy simples y otras más complicadas, unas rápidas y otras lentas, pero siempre obedecen a un esquema común: el virus reconoce su célula diana, entra en ella («la infecta»), se replica (produciendo mutaciones durante el proceso) en su interior, y su progenie sale (unas veces rompiendo la membrana celular y otras no) para infectar más células y continuar el ciclo hasta que no queden hospedadores disponibles. Por tanto, los virus son estructuras nanométricas capaces de infectar células y replicarse en ellas generando formas mutantes durante el proceso: son nanomáquinas que evolucionan. Este hecho los ha convertido en herramientas óptimas para ser utilizados en medicina, y más recientemente en aplicaciones nanobiotecnológicas.
Así, los virus o ciertas partes de ellos se han empleado por su potencial para fabricar vacunas que nos permiten prevenir las infecciones producidas por esos mismos virus o por otros similares. En la elaboración de vacunas obtenidas por ingeniería genética a partir de virus se suele emplear alguna de las proteínas de la cápsida o fragmentos de ellas, o bien el virus completo debidamente inactivado o «modificado» para que carezca de potencial replicativo y no produzca enfermedades en el paciente tratado. Otra aplicación médica relevante de los virus es aprovechar lo que la evolución ha puesto en nuestras manos: un enorme repertorio de nanomáquinas altamente especializadas, capaces de llegar a células concretas, entrar en ellas y destruirlas. En el apartado 7.5 se muestra cómo, para el desarrollo de algunas de las aplicaciones terapéuticas de los virus, sus propiedades mecánicas (rigidez y/o morfología de la cápsida) han de ser estudiadas en detalle, mediante una combinación de técnicas genéticas y nanotecnológicas. Por otra parte, como se describe en el apartado 7.10, se están diseñando virus modificados para que funcionen como nanodispensadores de fármacos en el organismo.
Además de los virus, existen muchas otras «nanomáquinas naturales» trabajando activamente en el interior de la célula. Entre ellas, quizá las más complejas e importantes son los ribosomas, responsables de traducir la información genética (el paso ARN→proteína): el ribosoma lee los nucleótidos del «ARN mensajero», y por cada tres de ellos une un aminoácido concreto a la proteína que se está sintetizando. Las reglas para traducir todos los posibles tripletes de cuatro nucleótidos del ARN (la combinatoria nos dice que son 43 = 64) a los 20 aminoácidos que forman las proteínas vienen fijadas en un diccionario común a todos los seres vivos: el denominado «código genético». Los ribosomas son partículas ribonucleoproteicas (es decir, formadas por ARN y proteínas) que existen en todos los tipos celulares de todas las especies. Tienen una forma redondeada sin simetría interna, un diámetro de entre 25 y 30 nm, y están constituidos por dos subunidades de diferente tamaño.
A lo largo del último medio siglo se ha utilizado un gran número de técnicas con objeto de profundizar en la estructura global del ribosoma, y con ello llegar a comprender el mecanismo de la traducción. Los dos métodos experimentales más empleados han sido la difracción de rayos X a partir de cristales de ribosomas, y la crio-microscopía electrónica seguida de reconstrucción tridimensional de imágenes. Con ello, durante los últimos quince años se han obtenido imágenes cristalográficas con una resolución de entre 0,1 y 0,2 nm, como se muestra en la Figura 7.4. Además, el uso combinado de varias técnicas estructurales ha permitido estudiar el movimiento coordinado de los componentes de estas complejas «nanofactorías de proteínas» durante el proceso de traducción.

FIGURA 7.4. Estructura cristalográfica de alta resolución de un ribosoma bacteriano durante la traducción del ARN mensajero. Se observan las subunidades ribosomales menor (izquierda, gris claro) y mayor (derecha, gris oscuro). El ARN mensajero se muestra como una cinta en la zona de interacción entre ambas subunidades, y la proteína que está siendo traducida corresponde a la hélice recta que avanza a través de la subunidad mayor hacia la parte inferior derecha de la figura. La barra corresponde a 10 nm. Imagen obtenida por el grupo de A. E. Yonath (Instituto Weizmann de Ciencias, Israel).
Otros ejemplos de nanomáquinas moleculares presentes en la célula (formadas principalmente por proteínas) son el replisoma (complejo de replicación del ADN), el complejo de transcripción del ADN a ARN, o el proteosoma (cuya función es la degradación controlada de proteínas). Todos ellos se están estudiando en profundidad desde hace décadas, y de su funcionamiento podrán extraerse claves para el diseño u optimización de diferentes nanomáquinas artificiales.
7.5. Manipulando biomoléculas individuales
Cada vez conocemos mejor los nanocomponentes del mundo vivo y sus interacciones. Además, los avances de la nanotecnología nos permiten hoy en día manejar biomoléculas de una en una para moverlas de un lugar a otro, entender cómo funcionan y qué tipo de contactos establecen entre sí. Esta posibilidad abre campos de investigación totalmente nuevos, ya que hasta hace pocos años los avances en biología molecular y biotecnología requerían el trabajo simultáneo con todas las biomoléculas presentes en una muestra compleja (como puede ser un fluido biológico) o pura (una preparación de laboratorio de un único tipo de moléculas, por ejemplo una proteína).
Con ello, se manejaban simultáneamente muchos billones de moléculas (diferentes o iguales) y por tanto lo estudiado era el comportamiento promedio de las mismas. Por el contrario, ahora ya es posible tomar una única molécula aislada (por ejemplo, una proteína o una cadena corta de ADN), un solo agregado macromolecular (por ejemplo un ribosoma), o un único virus entre los miles de millones que se producen cada día en un paciente infectado. Así, la nanotecnología permite estudiar el comportamiento de dicha entidad biológica individual y medir su respuesta frente a diversos estímulos.
Entre los sistemas desarrollados para manipular moléculas biológicas individuales, varios de ellos requieren la funcionalización previa de la biomolécula con otro tipo de molécula o con una nanoestructura artificial, como un nanotubo de carbono o una nanopartícula. Así, por ejemplo, si se une una nanopartícula a una cadena corta de ácido nucleico o a una proteína (gracias a la interacción con ambos de otra pequeña molécula que actúa como «puente»), el mayor tamaño y estabilidad de la nanopartícula permite manipular con más facilidad la biomolécula unida a ella dentro de la disolución que la contiene. Una de las formas más desarrolladas para mover y manipular biomoléculas individuales funcionalizadas con nanopartículas consiste en el empleo de un instrumento denominado «pinzas ópticas», también conocido como «trampa óptica». Este sistema, puesto a punto por A. Ashkin en los Laboratorios Bell en 1970, se basa en que un rayo láser altamente focalizado es capaz de atrapar y mover partículas dieléctricas (es decir, no conductoras de la electricidad) sin necesidad de «tocarlas» físicamente (Figura 7.5A).

FIGURA 7.5. Tecnologías para manipular biomoléculas individuales. A. Pinzas ópticas. B. AFM.
La alta sensibilidad de las pinzas ópticas permite realizar desplazamientos y rotaciones subnanométricos en objetos de tamaño comprendido entre aproximadamente 10 nm y 10 μm (entre ellos moléculas individuales, agregados macromoleculares, virus, orgánulos celulares o células completas), y cuantificar fuerzas muy pequeñas, en el rango del piconewton. Por tanto, este sistema permite investigar un gran número de procesos bioquímicos y biofísicos, estudiar las propiedades mecánicas de los biopolímeros y caracterizar el funcionamiento de las diferentes nanomáquinas y nanomotores moleculares que trabajan en el interior de las células.
Las pinzas ópticas se utilizan, por ejemplo, para estudiar las características mecánicas de los virus y de algunos de sus componentes. Entre ellos se ha investigado en profundidad el virus bacteriófago llamado Phi29, que infecta a la bacteria Bacillus subtilis. El genoma de este virus es una molécula de ADN de doble banda (ds ADN) de unos 19 300 pares de nucleótidos, cuya longitud es de aproximadamente 6,5 μm, es decir, casi siete veces más larga que la célula a la que infecta. Sin embargo, ese ADN está empaquetado en una cápsida viral, de forma icosaédrica, cuyo volumen es de solo 0,06 μm3. Al final del proceso infectivo, antes de salir de la célula y quedar listo para infectar a la siguiente, el virus Phi29 debe empaquetar su largo genoma en esa diminuta cápsida. Para ello resulta necesario que disponga de algún tipo de motor capaz de «tirar» de su ADN y empujarlo dentro de la cápsida. Dicho bionanomotor se localiza en el cuello o conector del virus (ver Figura 7.3A), y durante los últimos años se ha caracterizado a escala molecular: está formado por una serie de proteínas y moléculas de ARN que cambian coordinadamente de forma para realizar una fuerza que se traduce en un esfuerzo de tracción sobre el ADN viral hacia el interior de la cápsida (Figura 7.6).
Para conocer los detalles del funcionamiento de ese nanomotor y medir su potencia, C. Bustamante y su grupo de investigación en la Universidad de California en Berkeley (EE.UU.) lograron unir la molécula de ADN genómico de un Phi29 a una nanoparticula que podían sujetar mediante pinzas ópticas. El nanomotor del virus tiraba de un extremo de la molécula de ADN para introducirla en su cápsida (previamente inmovilizada a un soporte), y a la vez los investigadores tiraban de la nanopartícula unida al otro extremo. En expresión del propio Bustamante, era «como tirar de la cola de un gato que quería escaparse». Con ello se pudo determinar que el nanomotor de Phi29 realizaba una fuerza de aproximadamente 70 piconewtons para empaquetar su genoma. Aunque este número es muy pequeño en valor absoluto, en proporción al tamaño del virus equivale a una fuerza inmensa: si lo escaláramos a las dimensiones habituales en las máquinas construidas por los humanos, ese motor sería capaz de arrastrar seis aviones de carga. Entre las numerosas aplicaciones adicionales de las pinzas ópticas, es destacable su empleo para estudiar distintos aspectos relacionados con la dinámica de replicación del ADN, un campo en el que trabaja activamente el Centro Nacional de Biotecnología del CSIC en Madrid.
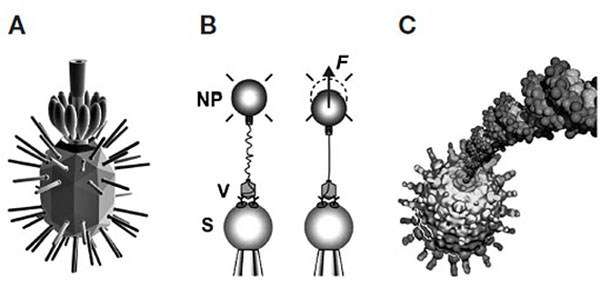
FIGURA 7.6. A. Recreación informática de la cápsida del bacteriófago Phi29, mostrando su nanomotor en el cuello del virus. B. Esquema del experimento realizado con pinzas ópticas para determinar la fuerza del nanomotor: S, soporte; V, virus; NP, nanopartícula; F, fuerza aplicada. C. Representación gráfica del nanomotor del virus empaquetando su genoma. Procedencia de las imágenes: A, K. Radcliffe y N. Stonehouse (Universidad de Leeds, Reino Unido); B y C, C. Bustamante (Universidad de Berkeley, California, Estados Unidos).
En paralelo al uso de pinzas ópticas, otro desarrollo nanotecnológico para manipular biomoléculas y complejos macromoleculares individualmente ha consistido en modificar la tecnología de AFM de forma que en la punta de la palanca se una (directamente, o a través de otra molécula que actúa como puente) uno de los extremos de la biomolécula estudiada. Con ello se dispone de una especie de «caña de pescar» (la micropalanca de AFM) provista de un «nanoanzuelo» (la punta de la palanca) en el que se ha unido un «nanocebo» (por ejemplo una proteína o un fragmento de ADN). Siguiendo con la metáfora, podemos «pescar» en una muestra biológica o solución donde exista la «presa» deseada (una molécula con afinidad por nuestro cebo) y medir la fuerza con la que debemos tirar de ella para capturarla. También se puede inmovilizar sobre un soporte el nanocebo por el extremo no unido al anzuelo y estudiar cómo cambia su estructura cuando tiramos de la caña. Con esta última variante se ha logrado, por ejemplo, cuantificar la fuerza necesaria para desplegar los distintos dominios estructurales en los que se organizan ciertas proteínas (Figura 7.5B). También se ha utilizado con éxito para estirar, comprimir, doblar, desenrollar o «superenrollar» (es decir, enrollar aún más) cadenas sencillas o dobles de ADN y de ARN, así como para hibridar y deshibridar de manera controlada las dos hebras complementarias de una doble hélice de ADN. La medida de la fuerza en cada caso nos informa sobre la elasticidad de las biomoléculas, y sobre el tipo de enlaces que mantienen su nanoestructura funcional.
La tecnología de AFM se está empleando también para profundizar en el conocimiento de la estructura de las partículas virales, y para estudiar sus características físicas como elasticidad y resistencia mecánica. En esta variante de la técnica, que se conoce como «espectroscopía de fuerzas», se presiona sobre la muestra (la cápsida del virus) con la punta de la micropalanca y se cuantifica la fuerza requerida para deformarla, aplastarla o romperla. Como ejemplo, investigadores de la Universidad Autónoma de Madrid y del Centro de Biología Molecular Severo Ochoa (CSIC-UAM) están estudiando el papel que desempeña el genoma de ADN del llamado «virus diminuto del ratón» en el mantenimiento de la estructura de la partícula viral. Para ello se analizan por separado partículas virales completas y cápsidas vacías, tanto en su variante natural como en el caso de que la cápsida esté constituida por proteínas del virus con determinadas mutaciones.
En el ámbito de la nanofluídica también se está trabajando con virus como sistemas modelo. Un buen ejemplo lo proporciona el virus del mosaico del tabaco, que tiene simetría helicoidal y forma de nanotubo con unos 300 nm de longitud, 18 nm de diámetro externo y 4 nm de diámetro interno (ver Figura 7.3B). Por tanto, constituye un modelo valioso para estudiar cómo fluyen los líquidos en tubos con diámetros menores de 30 nm, un límite a partir del cual se sabe muy poco sobre su comportamiento hidrodinámico y acerca de los métodos necesarios para lograr el confinamiento del fluido. Así, combinando estos nanotubos naturales con sistemas de micro— y nanofluídica, investigadores del CIC-Nanogune de San Sebastián están estudiando la difusión de iones metálicos (de hierro y níquel) y la migración de nanopartículas de oro por su interior, lo que se promueve depositando nanogotas mediante AFM en ambos extremos del virus.
7.6. El ADN como nanobiomaterial
Los ácidos nucleicos, debido a sus características estructurales, pueden ser utilizados como materiales muy útiles en bionanotecnología, en particular para construir distintos tipos de nanoestructuras. El ADN es una molécula polimérica formada por monómeros denominados desoxirribonucleótidos, cada uno de los cuales consta del azúcar desoxirribosa, un grupo fosfato, y una base nitrogenada (también llamada simplemente «base») que puede ser de cuatro tipos: adenina (A), guanina (G), citosina (C) y timina (T). En el otro ácido nucleico natural, el ARN, sus monómeros son ribonucleótidos formados por ribosa, el grupo fosfato, y cuatro tipos de bases: A, G, C y uracilo (U). La secuencia de bases de una molécula de ADN (por ejemplo, GATTACATACATACAAAG) constituye su mensaje genético. Por tanto, la información heredable almacenada en los genomas es la secuencia de sus nucleótidos: una larga «palabra» formada por miles de «letras» en los virus (por ejemplo, el de la hepatitis B tiene un genoma de ADN muy pequeño, con 3200 nucleótidos), millones en las bacterias (el ADN genómico de Escherichia coli posee 4 600 000 nucleótidos), o hasta miles de millones en las plantas y animales (el genoma de Homo sapiens es un largo texto con aproximadamente 3 300 000 000 letras).
Las técnicas de secuenciación o «lectura» de ácidos nucleicos, iniciadas en los años setenta, se basaban en la posibilidad de romper controladamente el ADN en ciertas posiciones, o bien en realizar la copia de una hebra forzando al sistema para que se detuviera en una posición concreta al introducir un «dideoxinucleótido terminador». El gran desarrollo y automatización del segundo de estos métodos permitió abordar distintos proyectos de secuenciación de genomas completos durante los años noventa. Así, por ejemplo utilizando esta técnica en decenas de laboratorios simultáneamente, el genoma de nuestra especie se completó casi en su totalidad en el año 2001. Continuando el progreso tecnológico, desde 2005 se dispone de sistemas de nueva generación para secuenciar ADN, con los cuales se ha logrado un enorme aumento en la velocidad de secuenciación y una reducción del coste por nucleótido secuenciado. En estos métodos la aportación de la nanotecnología ha sido fundamental para miniaturizar los dispositivos o compartimentos en los que se produce la reacción de secuenciación.
Como un paso más en esta carrera, los avances de la nanotecnología para manipular biomoléculas individuales (descritos en el apartado anterior) han originado sistemas «de tercera generación» con los que es posible secuenciar moléculas de ADN una a una. Así, la empresa británica Nanopore Technologies ha puesto en el mercado una plataforma que ofrece la posibilidad de secuenciar moléculas individuales de ADN (y también ARN o proteínas) analizando las alteraciones que se van produciendo en la diferencia de potencial eléctrico a uno y otro lado de una membrana artificial en la que se ha insertado un nanoporo formado por proteínas. El paso secuencial de los monómeros (los nucleótidos del ADN o ARN, o bien los aminoácidos de la proteína) a través de ese poro produce cambios de potencial transmembrana característicos para cada monómero, con lo que se va averiguando la secuencia de la molécula. El proceso está totalmente automatizado, y acoplado a los programas bioinformáticos que permiten interpretar el resultado de la secuenciación. En un desarrollo más reciente de este sistema se está investigando el uso de nanoporos formados por grafeno o por nitruro de silicio. Una característica relevante de los métodos de tercera generación es que permiten, por primera vez en la historia, secuenciar cualquier molécula de ADN sin que se requiera su amplificación previa utilizando métodos enzimáticos. Una nueva revolución en el campo de la genómica está servida.
En cuanto a su estructura, el ADN es una doble hélice formada por dos cadenas de nucleótidos cuyas bases nitrogenadas están orientadas hacia el eje de la hélice e interaccionan entre sí mediante puentes de hidrógeno. La interacción que se establece entre las bases obedece a la complementariedad A-T (cuando hay una A en una cadena, en la otra hay una T) y G-C. En su conformación más habitual el diámetro del ADN es de 2 nm, y la distancia entre los planos ocupados por dos bases consecutivas es de 0,34 nm. Dado que en cada vuelta completa de la doble hélice hay exactamente 10 nucleótidos, su «paso de rosca» es de 3,4 nm (Figura 7.7). El descubrimiento de la estructura y dimensiones del ADN se basó en las equivalencias de bases observadas por E. Chargaff, en los datos de difracción de rayos X obtenidos por R. Franklin y M. Wilkins, y en la síntesis de todas las evidencias experimentales que realizaron J. Watson y F. Crick en 1953.
La rigidez del ADN se ha podido estudiar recientemente empleando espectroscopía de fuerzas, una variante del AFM descrita en el apartado anterior. Así, se ha determinado que la longitud máxima que tiende a mantenerse extendida, sin plegarse o doblarse, es de aproximadamente 50 nm para el dsADN (lo que supone unos 150 nucleótidos) mientras que se reduce a 5 nm (15 nucleótidos) para el ssADN. Precisamente este último valor equivale a la longitud óptima de los oligonucleótidos de ssADN utilizados como sondas en microarrays y otros biosensores (ver apartado 7.8), pues previamente se había comprobado que el ADN de más de 18 nucleótidos tiene mayor tendencia a plegarse sobre sí mismo (lo que además se favorece si existen regiones con nucleótidos complementarios en su secuencia) y a no interaccionar correctamente con su ADN diana.

FIGURA 7.7. A. Dimensiones típicas de la doble hélice de ADN. B. Moléculas de dsADN visualizadas por AFM. Gentileza de R. M. Jáudenes y A. García-Sacristán (Centro de Astrobiología, CSIC-INTA).
En cuanto a su capacidad para almacenar información el ADN es un material extraordinario, con una densidad de información mucho mayor que la de los DVDs, discos duros y otros sistemas actualmente en uso (como comprobaremos en el siguiente párrafo). Además, el ADN resulta estable durante milenios (o incluso millones de años si las moléculas son cortas y sus condiciones de preservación han sido óptimas), y su información puede ser leída aunque las cadenas estén dañadas. A diferencia de los sistemas informáticos ideados por los humanos, el hardware para la lectura del ADN, formado por nucleótidos y enzimas, lleva miles de millones de años funcionando adecuadamente sin requerir ningún tipo de «actualización» o cambio de versión, y es totalmente compatible con cualquier «texto» de ADN.
Para demostrar la utilidad del ADN como sistema de almacenaje de información, investigadores de la Universidad de Harvard (EE. UU.) han codificado recientemente en forma de 55 000 oligonucleótidos (es decir, moléculas cortas de ADN) un libro formado por 53 426 palabras y 11 imágenes, y han sido capaces de leer sin problema dicha información. El libro había sido digitalizado en paquetes de 96 bits, convirtiendo cada dígito 0 en el nucleótido A o C, y los 1 en G o T. Con ello, se sintetizaron oligonucleótidos con 96 nucleótidos de datos, flanqueados por las secuencias necesarias para su amplificación enzimática in vitro, y para incluir una especie de «código de barras» que indicaba la posición de ese fragmento de información dentro del texto. Así, el libro entero cabe en una gota de disolución, y puede ser leído con la tecnología de amplificación y secuenciación disponible en cualquier laboratorio de biología molecular. Los investigadores han sido capaces de ensamblar y decodificar la información, y al compararla con el texto original han encontrando solo diez errores en sus 5,27 millones de bits. Además, este trabajo ha mostrado que un polímero tridimensional como el ADN puede empaquetar información con densidades cercanas a 1016 bits/mm3 frente a los aproximadamente 106 bits/mm3 de un soporte bidimensional como el DVD. Sin duda, las aplicaciones de este nanosistema biológico de almacenamiento de información pueden ser espectaculares.
Por otra parte, desde el punto de vista de su carga eléctrica la presencia de grupos fosfato a lo largo de la cadena de ADN le confiere una carga neta negativa a pH neutro. Por ello, muchos investigadores se han preguntado si la molécula de ADN podría ser utilizada como un conductor eléctrico. La nanotecnología está ayudando notablemente en este sentido, aunque los resultados obtenidos hasta la fecha son controvertidos. Así, algunos experimentos de AFM con una punta conductora han mostrado que la cadena sencilla no conduce la corriente eléctrica, mientras que la doble hélice sí lo hace. Sin embargo, otros resultados experimentales y ciertos cálculos teóricos cuestionan estas deducciones y proponen que el ADN no es conductor de la electricidad en ningún caso. Por tanto, en la actualidad aún no hay consenso sobre si será posible disponer de nanocables de ADN por los que pueda transportarse la corriente eléctrica.
Independientemente de su carga, dadas sus dimensiones el ADN es un nanobiopolímero con el cual es posible diseñar y construir nanoestructuras artificiales con diferentes formas y aplicaciones. Aprovechando que las hebras complementarias del ADN tienden a unirse como lo hacen los dos lados de una cremallera, es posible ensamblar cadenas de ADN con secuencias artificiales predefinidas (fácilmente sintetizables en los laboratorios) y construir así las bionanoestructuras tridimensionales que se desee. Por ejemplo, se han generado estructuras ramificadas de ADN con las cuales se aumenta mucho la capacidad de los biosensores para detectar secuencias concretas, como por ejemplo regiones del genoma de una bacteria o un virus presente en una muestra natural. En otros casos, se han diseñado cadenas de ADN parcialmente complementarias que se autoensamblan en poliedros regulares como tetraedros o cubos cuando se ponen en disolución (Figura 7.8A). Este sistema se conoce actualmente como «origami de ADN» por su similitud con el arte de origen japonés que en español conocemos como papiroflexia. Alardeando del dominio de esta técnica, científicos de la Universidad de Harvard (EE.UU.) han construido recientemente un alfabeto completo y una colección de símbolos, con «tamaño de fuente» de 100 nm, formados cada uno de ellos por cientos de moléculas de ADN de 42 nucleótidos capaces de autoensamblarse (Figura 7.8B). Otros investigadores de esa misma Universidad han diseñado un sistema modular para ensamblar moléculas de ADN de 34 nucleótidos y construir la estructura tridimensional que se desee, como si se tratase de un «nano-Lego» con infinitas posibilidades.

FIGURA 7.8. A. Fases del ensamblaje de un nanocubo de ADN (figura adaptada de N. C. Seeman, Nature, 2003). B. Alfabeto formado por moléculas de ADN, con «tamaño de fuente» de 100 nm, visualizadas por AFM (Instituto Wyss de Ingeniería bio-inspirada, Universidad de Harvard, EE.UU.). C. El arte del origami molecular: nanomatraz construido con hebras de ADN, y sus imágenes por TEM, barra de 50 nm (figura adaptada de D. Han, Science, 2011).
La similitud de esta versátil técnica con el origami es tal que se ha puesto a punto un sistema que permite diseñar no sólo poliedros regulares o formas con caras planas, sino también estructuras 3D curvadas como semiesferas, esferas completas, elipsoides e incluso «nanomatraces» que recuerdan a los que, con un tamaño un millón de veces mayor, se usan en los laboratorios (Figura 7.8C). El principal interés de estas nanoestructuras de ADN está en que pueden incluir otras moléculas en su interior, convirtiéndose en herramientas útiles en biomedicina. Así, se está explorando el uso de nanoestructuras de ADN como transportadores de fármacos o anticuerpos: estos nanocontenedores podrían entrar a una célula diana (por ejemplo tumoral o infectada por un virus), y en su interior la estructura de ADN se abriría controladamente (o bien sería degradada por las enzimas celulares) liberando su contenido activo.
Por último, además de utilizar el ADN como nanomaterial para construir estructuras y nanocontenedores, se han desarrollado métodos para unir zonas concretas del ADN a proteínas u otras biomoléculas, y para combinarlo con nanomateriales artificiales como nanopartículas de distinto tipo, dendrímeros, nanotubos de carbono, fullerenos o grafeno. De esta forma, dos cadenas complementarias de ADN pueden unirse a sendas moléculas o nanomateriales, que quedarán aproximados a la distancia deseada cuando las hebras hibriden entre sí. Así, por ejemplo, si una hebra de ADN inmovilizada sobre una superficie se hibrida a su cadena complementaria unida por un extremo a un nanotubo de carbono, se logra guiar el nanotubo hasta el punto concreto de la superficie en el que estaba anclada la primera hebra. Estos sistemas de funcionalización del ADN permiten también aumentar su capacidad para formar monocapas autoensambladas sobre diferentes materiales, y el desarrollo «capa a capa» de nuevos tipos de biosensores, de los que se hablará en el apartado 7.8.
7.7. Hacia la vida sintética
Dos de los ámbitos de investigación más fascinantes de la biología actual, a los que la nanotecnología también está contribuyendo, son el estudio del origen de la vida y la búsqueda de procedimientos para poder sintetizar vida artificial en los laboratorios. En ambos casos, una de las primeras dificultades que surgen es encontrar una definición de vida que permita distinguir claramente entre la materia inanimada y la viva, es decir, entre la química y la biología. Durante el período de unos 400 millones de años (Ma) en el que se originó la vida (entre hace 3900 Ma y hace 3500 Ma) se produjeron diferentes eventos moleculares que llevaron a la emergencia de sistemas compartimentados, con metabolismo y material genético capaces de replicarse y evolucionar por selección natural. Filósofos y científicos han situado el límite entre lo «vivo» y lo «no vivo» en distintas etapas de ese camino, generalmente en función de los intereses de su propio campo de investigación. Está claro que una roca amorfa no es un ser vivo, y que una bacteria sí lo es, pero ¿qué ocurre con los pasos intermedios que necesariamente debieron producirse? ¿Está vivo un cristal, una vesícula lipídica capaz de crecer y dividirse, o una molécula de ARN que tuviera capacidad para copiarse a sí misma?