15 лет ЧИП-секу
Биомолекула
🎈Что греха таить, по отдельности две технологии, которые «тщательно» спрятаны в названии метода, существовали давно. Но именно в ЧИП-секе они сошлись и составили мощную синергию. Давайте разобьём название метода на части и проведем небольшую дедукцию.
💾Во-первых, чип (от англ. CHromatin ImmunoPrecipitation) — иммунопреципитация хроматина. Его идея отнюдь не нова. Начиналось всё в 1969 году, когда появляются первые данные о том, что при обработке хроматина формальдегидом гистоны ковалентно сшиваются с ДНК. В 1984 г. Дэвид Гилмор и Джон Лис предложили уже собственно технологию иммунопреципитации для изучения ассоциации РНК-полимеразы с ДНК. Правда, сшивали они ультрафиолетом. Наконец, в статье 1988 г. в журнале Cell Марк Соломон и выходец из СССР Александр Варшавский окончательно зафиксировали классический протокол ЧИПа, применив и антитела, и формальдегидную сшивку.

🧵Какая же непосредственно последовательность ЧИПа? Сперва, разумеется, проводится обработка клеток сшивающим агентом, что индуцирует образование поперечных сшивок ДНК-белок. Затем — фрагментация ДНК на небольшие (до 1 тыс. п.н.) куски либо расщеплением микрококковой нуклеазой, либо обработкой ультразвуком (т.н. соникация). Хорошо, но где же в конце концов связь с иммунологией? Да вот она. После того как хроматин фрагментирован, для иммунопреципитации тандема белок-ДНК как раз используется подобранное антитело, специфичное к интересующему исследователя белку (подробнее про антитела). Между прочим, изготовление высокоспецифичных антител — один из самых трудоемких, даже лимитирующих этапов метода. Наконец, ковалентные сшивки разрывают нагревом, а ДНК, на которой сидели белочки интереса, очищается для дальнейшего анализа. Обязательно делается контроль — все те же процедуры с пропуском присоединения антитела.

🧬ДНК у нас в «кармане». Но что это за последовательности, мы не знаем. Как же их анализировать? Сначала это делали с помощью блоттинга, а потом ПЦР с геноспецифичными праймерами. Концентрацию фрагментов ДНК, содержащих интересующую область, сравнивали с концентрациями контроля.
🧠Но высокая производительность была достигнута, только когда началось использование гибридизации на ДНК-микрочипах. В конце 2000 г. в Science появляется публикация американских ученых во главе с Бингом Рэном, где и описывается новая стратегия анализа, которую нарекают… барабанная дробь…ЧИП-чипом! Ученые создали олигонуклеотидные микрочипы для полногеномного картирования сайтов присоединения таких трансфакторов как Gal4 и Ste12 у дрожжей. Флуоресцентно меченную ДНК гибридизовали с микрочипами и сравнивали с сигналом от контрольного ЧИПа или исходной ДНК. ЧИП-чип оказался хорош для дрожжей, но у него нашлись и ограничения: трудоемкость, сильный шум, неполный охват генома на микрочипе и проблема многочисленных повторов при анализе генома высших эукариот. Поэтому в 2007 г. «замучавшиеся» биологи нашли кое-что получше.

🖥Вот мы подобрались и ко второму компоненту — секу, то есть секвенированию, причем не простому, а массовому-параллельному (секвенирование тут). Да, именно NGS, появившееся еще в 2000-е, благодаря коммерциализации, к 2007 г. стало финансово доступнее. Кроме того, оказалось, что короткие фрагменты ДНК, выходящие из ЧИПа, идеальны по размеру для секвенаторов нового поколения. Так, за один прогон прибора типа Illumina стало возможно секвенировать миллионы фрагментов. Всё, что теперь оставалось после ЧИПа — отсеквенированные короткие фрагменты (риды), выровнять их на геном и на выходе получить пики. Пик — значит там и сидел наш гистон или трансфактор. Множество пиков складывается в очертание наподобие городской панорамы, анализ которой фактически и есть геномная аннотация. Безусловно, обработка данных ЧИП-сека — творческий процесс, требующий тесного сотрудничества между «мокрыми» биологами и биоинформатиками. Зато чувствительность такого метода вышла гораздо выше, чем у ЧИП-чипа, а также он независим от гибридизации. Кстати говоря, иммунопреципитация хроматина с последующим секвенированием стала одним из первых реальных применений NGS! Так зарождался ChIP-seq. Какие же были пионерские работы в столь перспективном направлении?
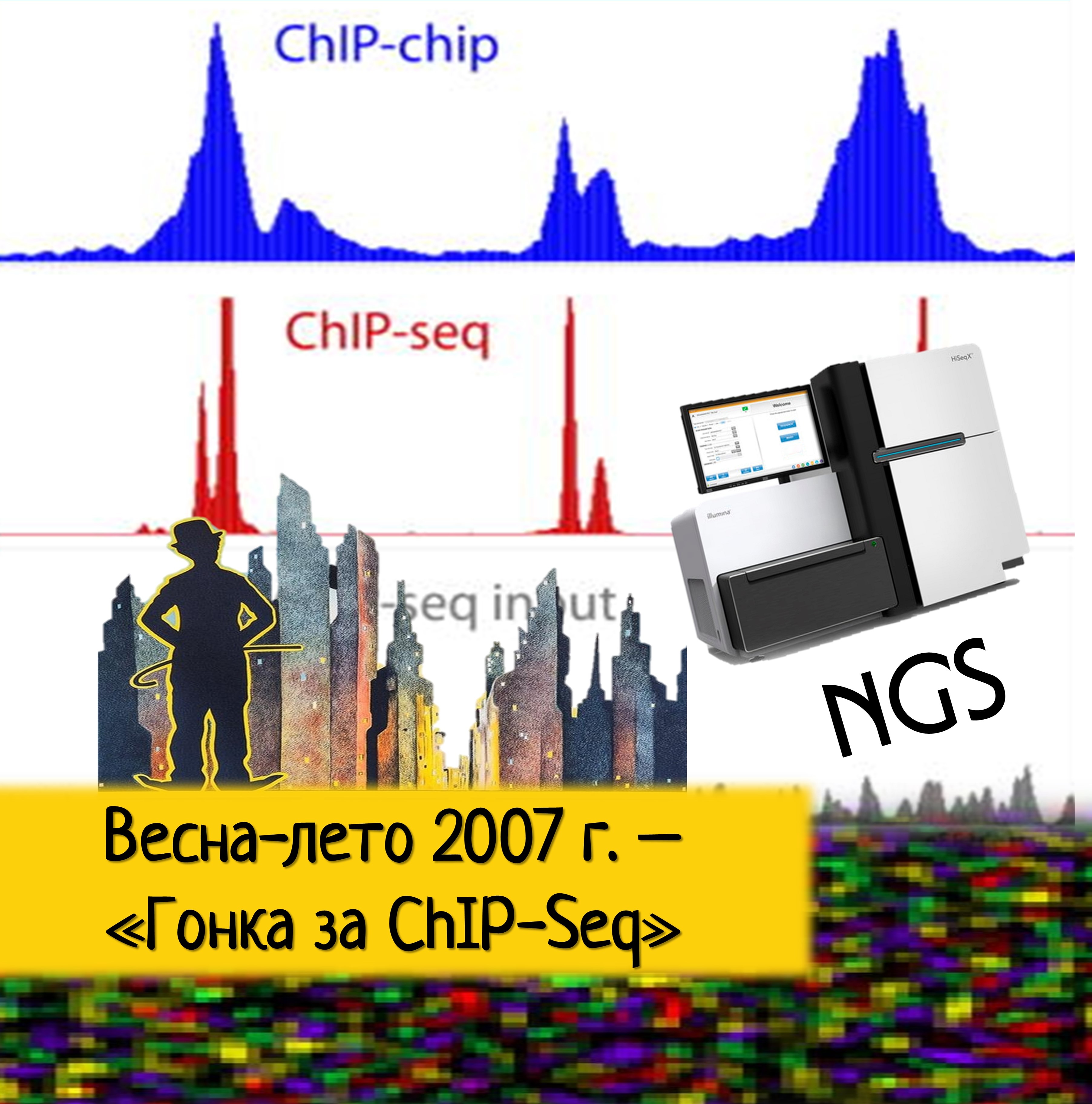
🧶Весной-летом 2007 г. одномоментно вышло несколько ключевых статей. Это можно назвать своеобразной гонкой за ЧИП-сек. Работы органично делились на 2 группы. Во-первых, это картирование модификаций белков-гистонов, верных спутников геномной ДНК. 18 мая 2007 г. в журнале Cell публикуется первая работа Артема Барского с соавторами из Национального института сердца, легких и крови в Бетесде Штата Мериленд в США. В Т-клетках человека ученые профилировали 20 модификаций гистонов, вариант гистона H2A.Z, а заодно еще РНК-полимеразу II и инсуляторный белок CTCF с охватом всего генома.
🐭Дальше эстафету подхватила группа Тарьея Миккельсена, Манчин Ку и товарищей. 1 июля выходит их статья в Nature. Биологи изучили триметилирование лизинов хвостов гистонов в эмбриональных стволовых клетках, фибробластах и клетках-предшественниках нейронов мышей. Интересно, что секвенирование позволило им различить аллельные варианты и даже проанализировать модификации хроматина в импринтированных областях!

🔗Вторым направлением стало картирование сайтов связывания транскрипционных факторов. 31 мая 2007 г. в Science вышла публикация смелых стэндфордских биологов Дэвида С. Джонсона, Барбары Волд (она, между прочим, научрук Ардема Патапутяна, который в 2021 получил Нобелевку по физмеду) и др. Они изучили связывание репрессора NRSF в клетках человека. NRSF имеет консенсусные сайты связывания в 21 п.н., что позволяет проводить биоинформатическое предсказание его мишени. ЧИП-сек подтвердил большинство таргетов, идентифицированных in silico!
🎡И, наконец, скажем о работе Робертсона и компании, с которой мы начали. 11 июня в журнале Nature Methods вышла его с соавторами статья, где помимо того, что провозглашалось открытие метода ChIP-seq, была и любопытнейшая содержательная составляющая. Канадцы проанализировали связывание компонента JAK/STAT-сигнального каскада трансфактора STAT1 в клетках HeLa и сравнили результаты с данными ЧИП-чипа. ЧИП-сек подтвердил 70% случаев, но показал высочайшую чувствительность, выявив почти в 4 раза больше сайтов!

🌺С 2010 г. настает расцвет ЧИП-сека, появляются многочисленные его модификации. В 2011 г., например, возникает ChIP-exo — когда после соникации применяют еще подрезку кончиков ДНК экзонуклеазой для повышения разрешения, а в 2014 г. рождается похожий X-ChIP-seq. Вообще традиционным недостатком подхода ChIP является невозможность различения факта связывания белка, происходящего во всей популяции, и события в одной клетке. Но и с этим справились: в 2015 г. возник single-cell ChIP-seq (недавно мы писали про одноклеточное РНК-секвенирование). Также в этом году появился ChIPmentation, когда адаптерные последовательности добавляются с помощью Tn5-транспозазы прямо к очищенным фрагментам хроматина, без необходимости сперва выделять ДНК. Потом ученым и вовсе надоело проводить подготовку хроматина, и они создали CUT&RUN и CUT&Tag (подробнее тут). А еще родной ЧИП-сек американские ученые умудрились «породнить» с CRISPR/Cas9…

🧫Надо сказать, крутизну ЧИП-сека поняли сразу. Крупные консорциумы, такие как ENCODE, «Дорожная карта эпигеномного картирования», modENCODE оказались одними из первых, кто использовал этот инструмент для характеристики эпигеномных профилей клеток, тканей организма и популярных клеточных линий. Быстрое и широкое внедрение ЧИП-сека в различных областях, таких как генетика развития, эволюционная биология и исследования эпигенетических вариантов рака, послужило основой для качественного скачка в понимании регуляторных сетей транскрипции, дифференциальной регуляции генов, функционирования хроматина.

А еще, помимо всего прочего, ЧИП-сек помог сделать «цветовую» классификацию хроматина плодовой мушки дрозофилы и других модельных объектов (мастрид).
